Das Forschungskonsortium leistet einen Beitrag zur digitalen Transformation des Trinkwassersektors, indem zwei Software Tools zur kontinuierlichen Überwachung (I) und Simulation (II) organischer und mikrobieller Trinkwasserqualitätsparameter entwickelt bzw. weiterentwickelt wurden. Das Projekt MoDiCon wurde im Rahmen der Deutsch-Israelischen Wassertechnologie-Kooperation im Call von 2019 durchgeführt (Förderkennzeichen: 02WIL1553).
An der Entwicklung der Software-Tools waren drei Forschungsgruppen der Technischen Universität Hamburg, des „Technion – Israel Institute of Technology“ und der Technischen Universität Ilmenau in Zusammenarbeit mit industriellen Partnern beteiligt. Die entwickelten Software-Tools ermöglichen eine kontinuierliche Beurteilung der aktuellen und zukünftigen Trinkwasserqualität im Trinkwassernetzwerk, was im Hinblick auf mikrobiologische Risiken eine Innovation darstellt. Im Rahmen des Projekts wurden zwei Software-Tools entwickelt bzw. weiterentwickelt, die zur Überwachung und Modellierung von Wasserqualitätsparametern direkt angewandt werden können:
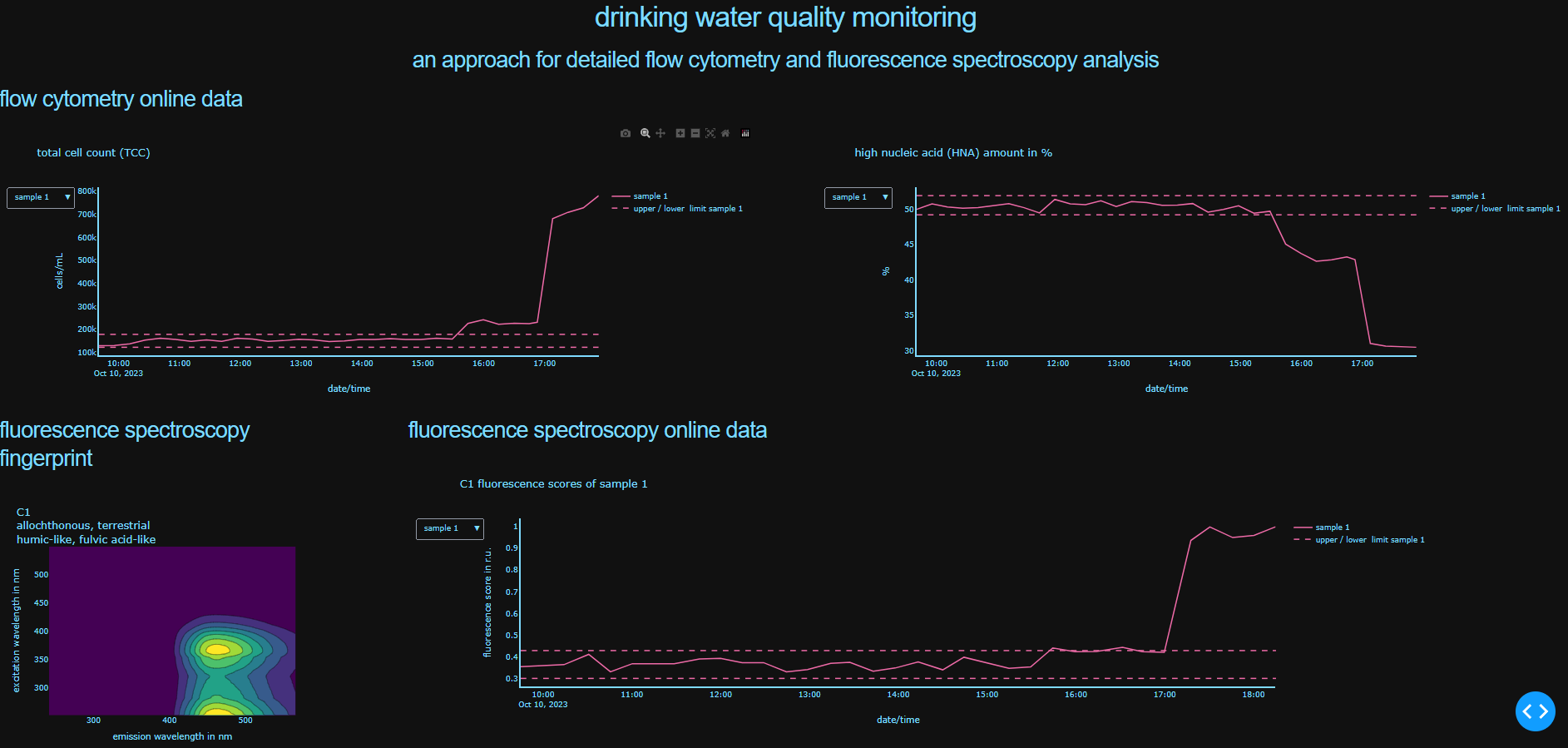
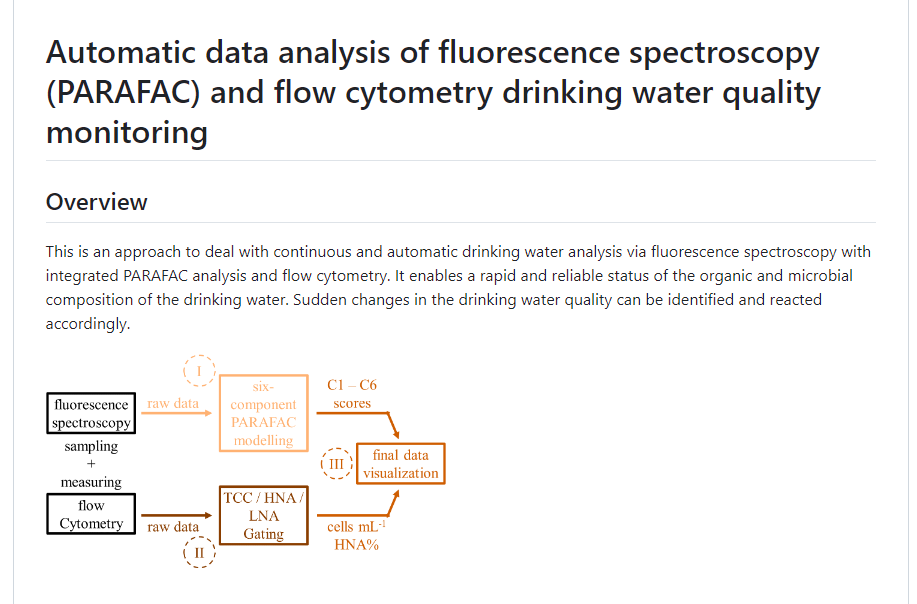
1. "Automized data analysis of fluorescence spectroscopy (PARAFAC) and flow cytometry drinking water quality monitoring"
Der entwickelte Ansatz ermöglicht die vollständig automatisierte parallele Auswertung und Visualisierung von kontinuierlich gemessenen Trinkwasserproben mittels Fluoreszenzspektroskopie und Durchflusszytometrie. Dabei wird die etablierte PARAFAC-Analyse in einer automatisierten Form angewandt, um organische Substanzen im untersuchten Wasser durch sechs organische Fluoreszenzkomponenten darzustellen. Außerdem wird das Wasser durch die Zellzahl sowie den Anteil an Zellen mit relativ hohen Nukleinsäuregehalt (HNA) charakterisiert. Durch die visuelle Darstellung in einem interaktiven Dashboard kann so eine Veränderung der Trinkwasserqualität in beinahe Realzeit (15 min) umgesetzt werden.
Auf das Software-Tool kann in der Mediathek unter „Automatisierte Datenanalyse“ zugegriffen werden.
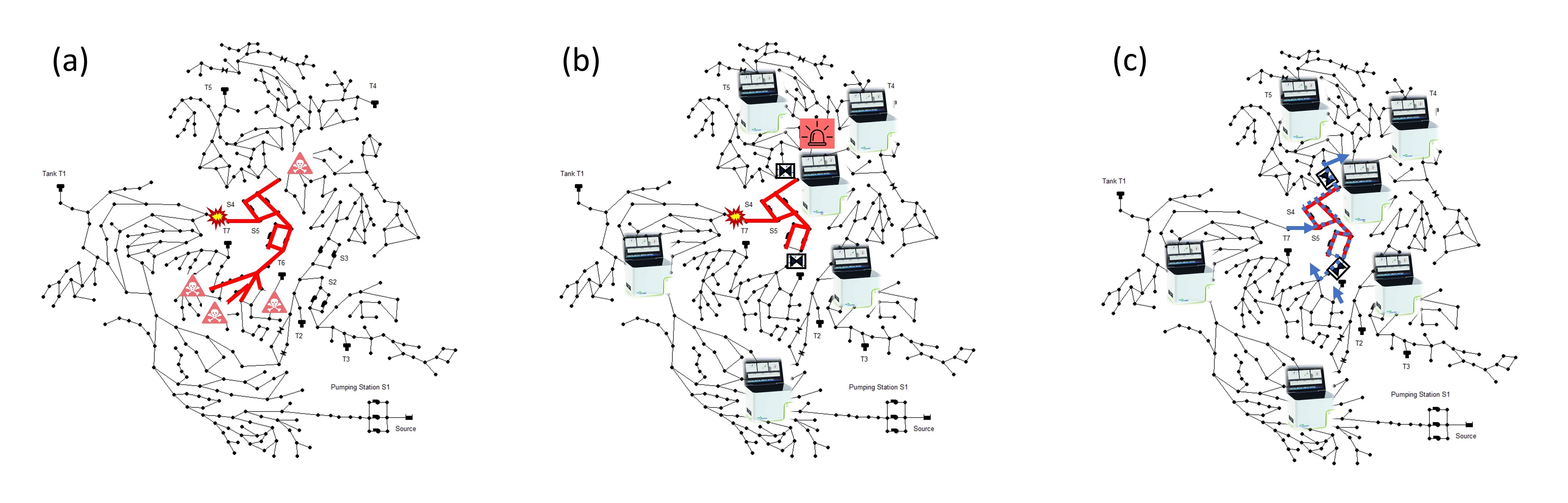
2. "EPyT-C – Fully independent multi species reactive transport modelling extension for EPyT (EPANET-Python)"
Die Erweiterung "EPyT-C" enthält die Möglichkeit zur Modellierung des reaktiven Transports mehrerer Substanzen (u.a. Schadstoffe), auch bekannt als multi-species reactive-transport (MSRT)-Modellierung. Explizit bietet dies mittels zwei entwickelter Module zum einen die Möglichkeit zur Modellierung des freien Chlors sowie der damit in Verbindung stehenden Konzentration an Desinfektionsnebenprodukten wie Trihalomethanen. Das zweite Modul ermöglicht die Modellierung der Wiederverkeimung im Trinkwassernetzwerk anhand u.a. der Konzentration lebender Bakterien.
Auf das Software-Tool kann in der Mediathek über den Beitrag „EPyT (EPANET-Python) - Unabhängige Multi-Spezies-Modellierung des reaktiven Transports“ zugegriffen werden.
Beide Software-Tools wurden an der Technischen Universität Hamburg und dem Technion erfolgreich im Labor- und Pilotmaßstab validiert und sind via GitHub (siehe Mediathek) als Open-Access Software frei verfügbar. Die öffentliche Verfügbarkeit ermöglicht:
· Die direkte Implementierung in vorhandene Trinkwassernetzwerke mit entsprechender Analytik.
· Die Möglichkeit zur individuellen Anpassung bzw. Erweiterung durch entsprechendes Fachpersonal.